4月6日,上海科技大学免疫化学研究所研究员杨海涛与清华大学教授、上海科技大学免疫化学研究所特聘教授、中国科学院院士饶子和团队在国际著名期刊《美国科学院院刊》(PNAS)在线发表研究论文,报道了新型冠状病毒的关键药靶——主蛋白酶在静息态、结合底物中间态和切割后状态的高分辨率晶体结构,以拍“全息照片”的方式精确地揭示了主蛋白酶催化底物水解的整个过程。同时,研究人员还发现新冠病毒主蛋白酶在识别病毒复制酶多蛋白过程中的关键特性:它能识别10个氨基酸残基长度的底物,但只对其中4个位点具有选择性。
自2019年12月以来,新冠疫情暴发并且迅速席卷全球。新冠病毒属于β冠状病毒属,是一种正链RNA病毒。新冠病毒的一大特征是当病毒入侵宿主细胞后,首先需要利用宿主的翻译机器将病毒遗传物质翻译成多聚蛋白(polyprotein)。其中两条超长的复制酶多蛋白(pp1a和pp1ab)经过病毒自身的两个蛋白水解酶主蛋白酶(Mpro)和木瓜蛋白酶(PLpro)精确水解切割,得以释放成熟的非结构蛋白(nsp 1-16)。这些非结构蛋白的一部分参与病毒转录复制复合体(RTC)的组装,启动病毒复制。主蛋白酶负责至少11个位点的切割,对新冠病毒的成熟和增殖起着至关重要的作用,而人体内没有类似的蛋白,所以主蛋白酶成为了抗病毒药物研发的关键靶点。
作为关键的药物靶点,新冠病毒主蛋白酶如何精确识别和切割复制酶蛋白的分子机制尚有待解答。为了探究这个问题,研究团队先后解析了新冠病毒主蛋白酶在静息态、结合底物中间态和切割后状态的高分辨率晶体结构(图1),揭示了主蛋白酶在整个催化反应过程中的三维空间构象的精细变化。同时,新冠病毒主蛋白酶在长达7000多个氨基酸的复制酶多蛋白上至少包含了11个切割位点,为了进一步探究主蛋白酶如何特异性的识别这些位点,研究团队分别解析了主蛋白酶突变体与6个位点的多肽底物的高分辨率复合物结构(图2)。研究分析发现新冠病毒主蛋白酶能识别10个氨基酸残基长度的底物,但只对其中S1、S2、S4和S1′这4个位点具有选择性。该项研究首次系统地报道了新冠病毒主蛋白酶的底物识别特异性和催化机制,为设计更加高效的抗新冠病毒药物奠定了重要的结构基础。
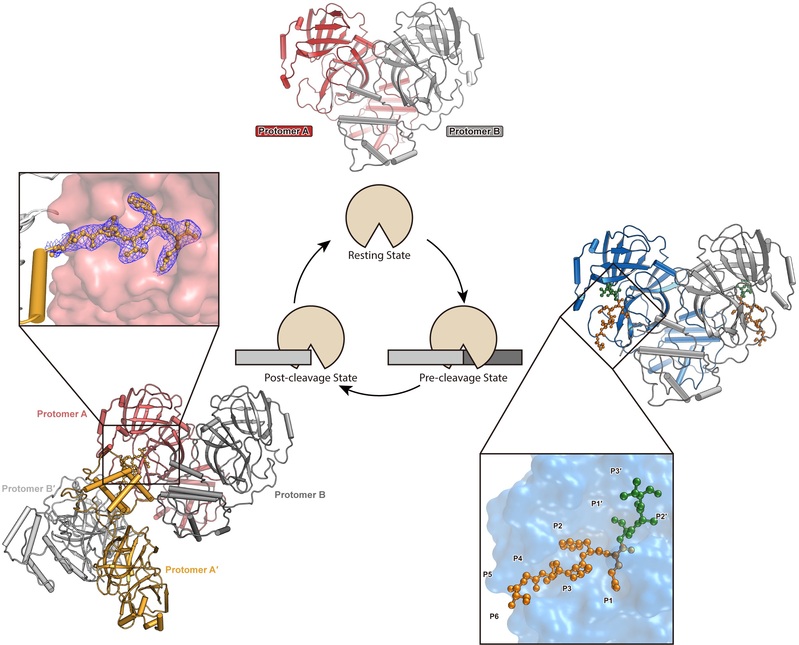
图1. 新冠病毒主蛋白酶在静息态、结合底物中间态状和切割后状态的高分辨率晶体结构
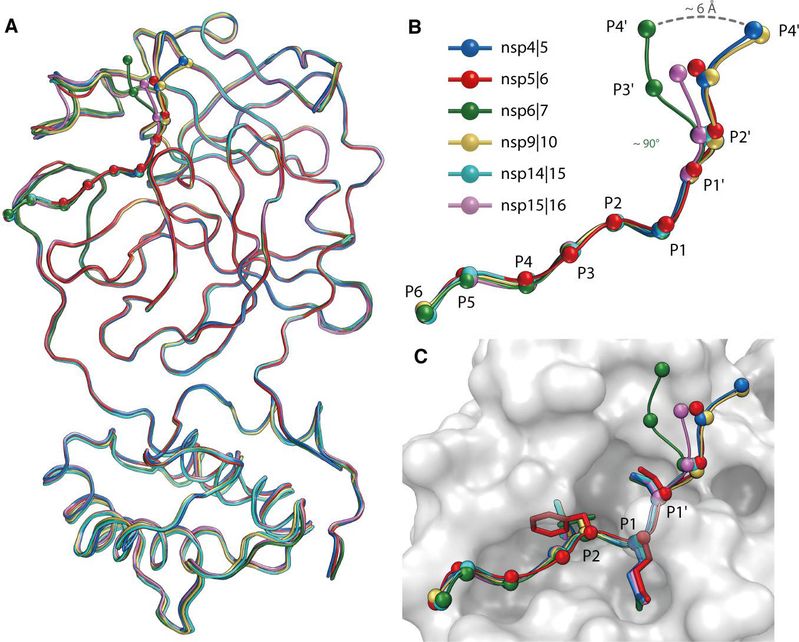
图2. 新冠病毒主蛋白酶识别底物的共性与差异
免化所抗结核结构研究中心长期致力于新发、再发传染性疾病病原体的结构生物学研究及新药开发,这是该中心在新冠攻关的又一重要研究成果。免化所副研究员赵耀、免化所与生命学院博士研究生朱艳、南开大学副研究员刘祥为本文的共同第一作者。杨海涛、饶子和和赵耀为本文的共同通讯作者。上海科技大学为第一完成单位。免化所副研究员张兵、副研究员杨秀娜,iHuman研究所武丽杰,美国克利夫兰诊所杨楷林也参与了此项研究。
论文标题:Structural basis for replicase polyprotein cleavage and substrate specificity of main protease from SARS-CoV-2



