近日,上海科技大学免疫化学研究所白芳课题组开发了基于蛋白质-蛋白质识别基序(motif)信息,挖掘新蛋白质-蛋白质互作体系并预测复合物三维结构新计算方法PPI-Miner,该工作发表于美国化学会Journal of Chemical Information and Modeling杂志(https://doi.org/10.1021/acs.jcim.2c01033),题目为“PPI-Miner: A Structure and Sequence Motif Co-Driven Protein-Protein Interaction Mining and Modeling Computational Method”。方法以网络服务器形式在课题组实验室网站对外开放(https://bailab.siais.shanghaitech.edu.cn/services/ppi-miner)。该方法以用户提供的蛋白质识别三维结构基序或者二维序列信息基序作为提问片段,通过遍历课题组构建的近乎覆盖全人类基因组的高质量蛋白质结构数据库,以挖掘候选互作蛋白质互作对,同时,该方法可用于发掘和鉴定特定E3水解酶可降解靶标,为分子胶水设计提供候选底物靶标,该方法已提供1,739种被热门分子胶水靶点CRBN所识别的潜在底物蛋白,并在线公开( https://bailab.siais.shanghaitech.edu.cn/services/crbn-subslib),该部分工作得到了上科大生命科学与技术学院仓勇教授的大力支持。
蛋白质-蛋白质相互作用是细胞生命活动的重要组成部分,在细胞的信号转导、细胞结构的组装、病原体-宿主的识别等重要生命过程中起到不可或缺的作用。目前,已经有多种策略用于蛋白质-蛋白质相互作用的识别和建模,其主要思路有:序列比对、共进化分析、蛋白-蛋白对接、相互作用网络分析以及一些多方法联合策略等。近年来,AlphaFold和RoseTTAFold的出现更进一步推动了蛋白质-蛋白质相互作用发现的方法学进步。在本项研究中,研究者发现在蛋白质-蛋白质相互作用界面上存在一些高度保守的结构片段,这些片段往往具有序列保守性或者是骨架结构保守性(图1),在多种蛋白与相同受体的复合物结构中呈现出非常保守的结合模式。那么,我们能否利用这些局部片段的保守性来挖掘新的蛋白质-蛋白质相互作用呢?

图1.两种蛋白质-蛋白质相互作用基序示例
为了得到这一问题的答案,课题组开发了两套用于挖掘人类蛋白质组中潜在序列基序或者结构基序的系统,分别命名为2DPPI-Miner和3DPPI-Miner。首先,研究者建立了这两种PPI挖掘系统所需要的蛋白质序列和结构数据库,两个数据库分别对应于人类蛋白质组的无序区域和有序区域,以实现对人类蛋白质组绝大部分区域的覆盖。随后,研究者首先开发并测试了2DPPI-Miner方法,值得注意的是,在本项工作发表之前,通过序列比对发现新的蛋白质-蛋白质相互作用对就已经是一种经典的研究思路,其中,使用BLOSUM矩阵进行序列相似性评估以排序搜索到的蛋白序列是一种主流的方法。在本项工作中,研究者采用ΔΔG结合自由能计算的方式来对搜索到的基序进行排序,结果显示,在用于测试的三个靶标上,2DPPI-Miner均优于基于序列相似性的评估方法。
除了传统的序列基序以外,研究团队还考虑到了序列不保守而骨架结构保守的结构基序,这一类型的基序往往在一些较为复杂的蛋白质-蛋白质相互作用界面上被发现,其中的典型代表是热门的分子胶水靶点CRBN,其底物识别基序是一段结构上保守但序列非常多样的β-hairpin结构。研究团队利用目前已知的所有CRBN底物以及其结合基序,评测了2DPPI-Miner和3DPPI-Miner模式在鉴定CRBN的正确结合基序上的表现,其中3DPPI-Miner表现出了最佳的早期富集能力和AUROC。值得注意的是,3DPPI-Miner中的碰撞过滤器除了可以提高3DPPI-Miner识别正确基序的能力,还可以快速清除一些发生碰撞的不合理结构,从而大大加快了整个方法的运行速度。
基于上述的两种PPI-Miner方法,课题组进一步在人类蛋白质组中进行了CRBN底物的搜索,结果显示,在搜索到的1,739种底物蛋白中,3DPPI-Miner不仅成功复现了大部分的已经解析的CRBN-底物复合物结构(包括IKZF1,SALL4,KC1A,ERF3A等,如图2A),还成功鉴定出了一些被其他研究团队通过实验手段发现的新型CRBN底物(如图2B)。此外,研究团队还发现有1,090种底物缺乏对应的ChEBI条目,且同时存在与CRBN的亚细胞共定位,这些蛋白可以作为接下来CRBN分子胶开发的靶标。
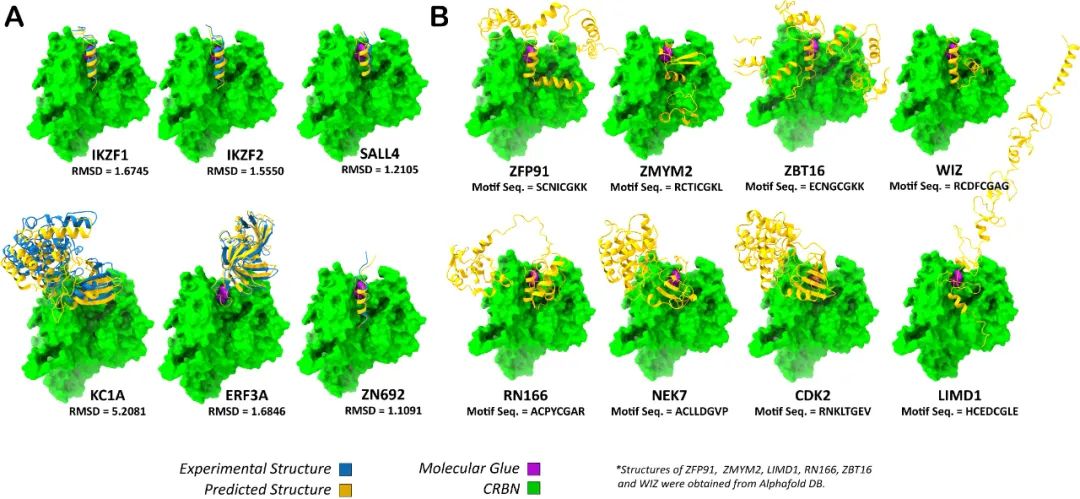
图2.CRBN潜在底物库的搜索
PPI-Miner建立了一套基于已知蛋白质基序搜索新的潜在蛋白质-蛋白质相互作用伙伴的完整方法学,这一方法既可以被用于新蛋白质-蛋白质相互作用发现,也可以作为蛋白质设计中蛋白质支架搜索的一种解决方案。未来,PPI-Miner有望帮助研究者发现更多潜在的蛋白质-蛋白质相互作用,为分子胶水的理性设计、新药物靶标的发现等领域提供新的动力。目前,这项研究的数据库及代码已经在代码托管平台GitHub开源(https://github.com/Wang-Lin-boop/PPI-Miner),值得注意的是,研究团队也开源了其数据库以及构建代码,以供国内外同行复现其工作以及构建其他物种的用于PPI-Miner方法的数据库。
免化所2021级博士研究生王林为论文第一作者,白芳研究员为论文的通讯作者。上海科技大学为第一完成单位。该项工作得到了科技部重点研发计划、临港实验室、国家自然科学基金委、上海市科委和上海市教委项目(上海市生物大分子与精准医药前沿科学研究基地等)的支持。该项工作也得到上科大高性能计算平台的全力支持。
原文链接:https://doi.org/10.1021/acs.jcim.2c01033



