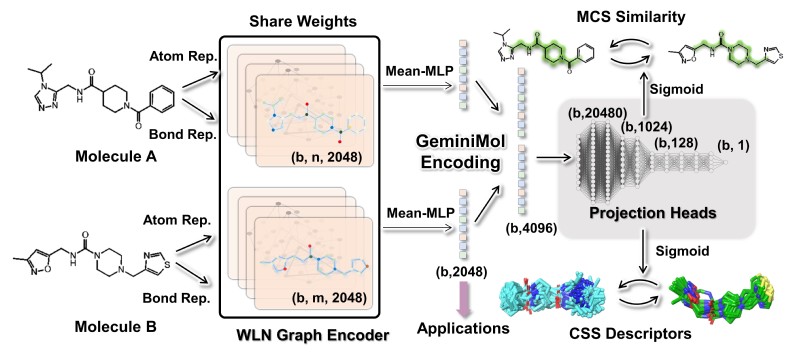
基于此,该研究设计了如图1所示的考虑分子三维构象空间的分子对比学习表征预训练模型。在预训练过程中,一对药物小分子首先通过相同的分子编码器进行独立的编码,得到一个2048维的分子表征向量,随后,使用多个不同的预测头将两个分子的表征向量投影到多种分子间相似性指标,包括分子的二维最大公共子结构相似性和分子的三维构象空间相似性(通过具有分子构象信息的药效团形状相似性来计算)。
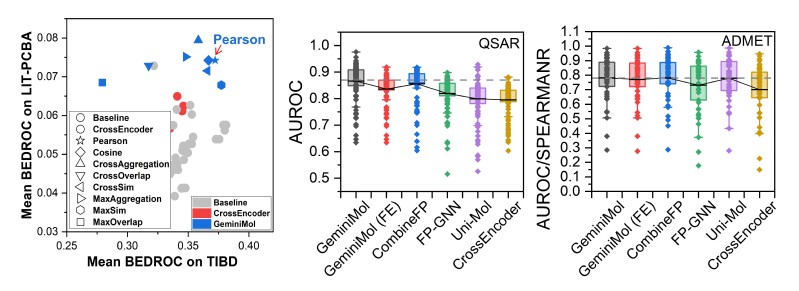
随后,研究团队在多种下游任务上对GeminiMol模型进行了基准测试,包括来自高通量筛选实验数据的虚拟筛选基准测试集LIT-PCBA、来自药物-靶标相互作用数据的靶标鉴定基准测试集TIBD、以及多种来源的定量构效关系(QSAR)和药物属性(ADMET)基准数据集。如图2所示,GeminiMol在多种药物发现下游任务上表现出较为均衡的优良性能,进一步证实了它应用于多种药物发现任务的优良潜力。该方法已在课题组多个药物研发应用项目中起到关键的推动作用。
论文的所有训练数据集、下游任务基准测试集、药物筛选应用所需的化合物数据集均已经开放储存在Zenodo仓库(https://zenodo.org/records/10450788),模型上传至HuggingFace(https://huggingface.co/AlphaMWang/GeminiMol),GeminiMol模型代码、分子指纹基线方法和使用教程均已开源在GitHub仓库(https://github.com/Wang-Lin-boop/GeminiMol)。
上海科技大学生命学院/免化所2024届博士毕业生王林(现为苏州系统医学研究所博士后)为本文的第一作者,免化所研究员、生命学院助理教授白芳为本文的通讯作者。另外,生命学院与信息学院多名研究生或者本科生也参与了本研究工作。上海科技大学为唯一完成单位。
论文链接:
https://onlinelibrary.wiley.com/doi/10.1002/advs.202403998



