11月29日,上海科技大学免疫化学研究所张贺桥/Roger Kornberg研究团队(结构生物化学课题组)在国际学术期刊《自然-通讯》(Nature Communications)在线发表了题为“Mechanistic insights into histone recognition and H3K14 acetylation by the NuA3 histone acetyltransferase complex” 的研究论文,报道了真核生物组蛋白乙酰转移酶复合物NuA3与底物组蛋白H3尾部结合并特异性乙酰化H3K14位点的结构特征。

真核生物的遗传物质以核小体为基本结构单元,每个核小体由一个组蛋白八聚体(包括H2A、H2B、H3 和 H4)以及其外围缠绕约 147 bp 的 DNA 所组成。组蛋白 N 端尾部的特定赖氨酸和精氨酸残基可发生多种翻译后修饰,如甲基化、乙酰化、磷酸化和泛素化等。这些修饰及其介导的生物学效应被表观遗传学先驱 C. David Allis 称为“组蛋白密码”(histone code),对基因表达调控具有至关重要的作用。研究表明,真核生物中存在多种负责添加或去除这些修饰的“书写器”(writers)和“擦除器”(erasers)。其中,NuA3 复合物是催化组蛋白 H3K14 乙酰化的重要“书写器”之一。H3K14乙酰化是调控染色质开放性和基因转录激活的重要标记,在基因表达、细胞分化和DNA修复中发挥关键作用。此前的结构生物学研究多集中于催化亚基的MYST结构域,而该复合物的整体结构及其对底物H3K14识别和乙酰化的分子机制一直未被解析。
本研究利用昆虫细胞表达系统成功重组并纯化出了高纯度、高均一性的NuA3复合物,并在体外证实其对H3K14的特异性乙酰化活性。随后采用单颗粒冷冻电镜技术,分别解析了NuA3在未结合底物状态、结合乙酰辅酶A的状态,以及同时结合组蛋白H3尾部和乙酰辅酶A的复合物状态的结构。研究显示,Sas3和Nto1亚基协同构成了NuA3的核心框架,为复合物的稳定性和底物识别提供关键支撑(图1A)。
NuA3的组蛋白尾部结合裂隙由催化亚基Sas3与非催化亚基Nto1共同构成(图1B,1C),显示了非催化亚基对于底物识别的重要作用。该裂隙通过疏水作用识别H3第9–12位残基,并通过极性相互作用识别第13–15位残基。对多个关键氨基酸位点进行的突变与活性分析验证了这些相互作用在催化过程中的重要性。值得注意的是,H3K14前的Gly13因其侧链体积小,能够避免与结合裂隙中的氨基酸发生空间冲突。因此,Gly13对底物特异性具有关键作用(图1C)。研究显示:NuA3负责催化的氨基酸Glu452、Cys418,组蛋白H3K14(突变为甲硫氨酸)、乙酰辅酶A的乙酰基团三者处于预反应状态(图1D)。结构比对还表明,乙酰辅酶A的结合会诱导“组蛋白结合环”和“CoA结合螺旋”发生构象变化,从而扩大结合裂隙,为组蛋白尾部进入结合位点和后续反应做好准备(图1E)。
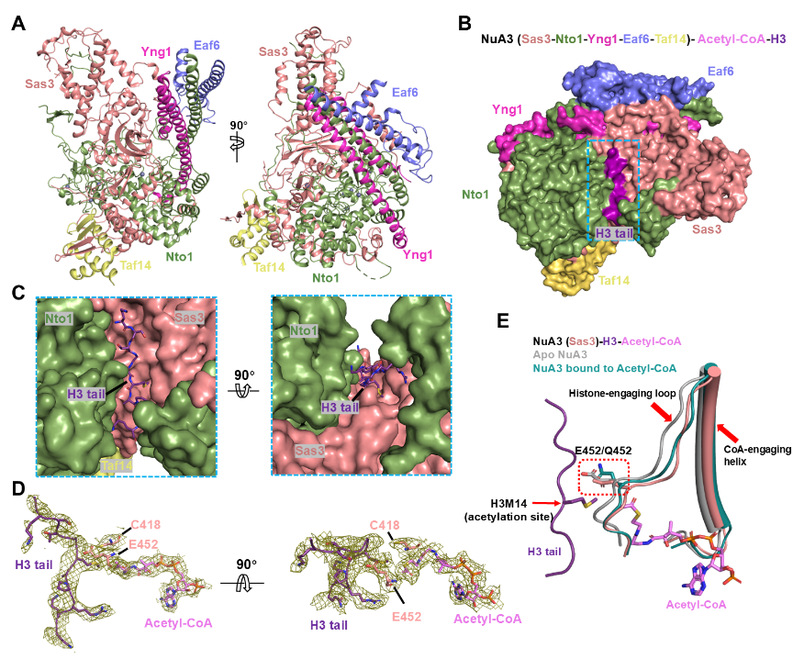
图1. NuA3复合物识别和乙酰化组蛋白H3K14分子机制。(A)NuA3复合物冷冻电镜结构;(B)NuA3与组蛋白H3尾部及乙酰辅酶A结合后的复合物结构;(C) 组蛋白H3尾部在NuA3底物结合裂隙中的放大视图;(D) 组蛋白H3尾部、乙酰辅酶A及NuA3催化氨基酸局部电子密度图;(E)乙酰辅酶A和H3尾部结合诱导NuA3发生构象变化
本研究还将该机制进一步拓展至人类同源复合物BRPF1/2–HBO1–ING5–MEAF6,结果表明其组蛋白识别机制与酵母NuA3具有高度保守性,为后续开展更广泛的表观遗传调控研究奠定了结构基础。
免化所结构生物化学课题组2025级博士研究生史雯萍、高级工程师赵莉霞,以及2024级博士研究生王逸儒为本研究共同第一作者。结构生物化学课题组负责人张贺桥副研究员、Roger Kornberg教授为本论文共同通讯作者。上海科技大学为论文第一完成单位。
论文链接:https://www.nature.com/articles/s41467-025-67049-0



